Protéine du rétinoblastome
La protéine du rétinoblastome est une protéine de séquestration qui exerce un contrôle négatif du cycle cellulaire. Cette fonction est principale dans les organismes pluricellulaires pour éviter la formation de tumeurs malignes qui mettraient en péril l'organisme,...
La protéine du rétinoblastome (pRB) est une protéine de séquestration qui exerce un contrôle négatif du cycle cellulaire. Cette fonction est principale dans les organismes pluricellulaires pour éviter la formation de tumeurs malignes qui mettraient en péril l'organisme, ce qui sert à qualifier cette protéine de «suppresseur de tumeur».
Son nom vient de son étroite collaboration dans un cancer ophtalmologique pédiatrique : le rétinoblastome. Cette protéine monomérique a un poids moléculaire de 105 kilodaltons, est plutôt instable (index d'instabilité in vitro de 47, 85) et est hydrophile (hydropathie de -0, 406).
Gène
Chez l'homme, le gène codant pRB est le gène RB1. Ce gène est localisé sur le chromosome 13 à l'emplacement 13q14.2. Ce gène est particulièrement long et contient beaucoup d'introns et peu de séquences codantes.
Structure
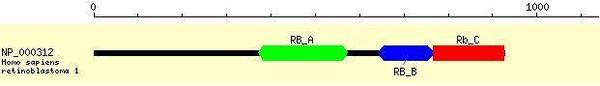
Chez l'homme pRB est une protéine de 928 acides aminés et comporte 3 domaines[1], [2] :
- pRB_A : Site de liaison de la cycline,
- pRB_B : Domaine de liaison. Site d'interaction avec des protéines virales,
- pRB_C : Domaine carboxyl-terminal. Site de liaison requis pour lier la protéine E2F. Site de phosphorylation des CDK4 et CDK6.
Les mutations affectant la fonction biologique de pRB sont le plus souvent des mutations ponctuelles d'une seule paire de bases menant à une mutation non sens. [3] Ces mutations empêchent la protéine de séquestrer correctement le complexe E2F/DP. Si ce complexe n'est pas correctement séquestré, le facteur de transcription E2F peut toujours faire continuer le cycle cellulaire. Puisque pRB est une protéine monomérique, il est indispensable d'avoir les 2 allèles du gène RB1 muté pour voir se manifester la condition pathologique.
Fonction biologique
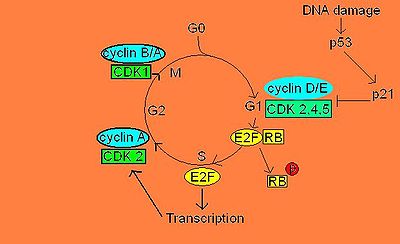
pRB a un mécanisme de suppression tumorale, qui bloque le cycle cellulaire en phase G1 via la séquestration du facteur de transcription E2F.
Ce «frein» moléculaire est essentiel, car sans lui, la cellule serait toujours en division cellulaire. Dans la majorité des cellules somatiques d'un organisme adulte, pRB n'est pas phosphorylée, car les cellules de l'organisme adulte n'ont presque jamais besoin de se diviser. pRB n'étant pas phosphorylée, la libération du complexe E2F/DP est inhibée. Cette inhibition stoppe le cycle cellulaire en phase G1. Quand la cellule est en division active, des complexes cycline/CDK (cyclin dependant kinases) viennent phosphoryler pRB au niveau du domaine C. Cette phosphorylation amène un changement de conformation protéique donnant la possibilité le relâchement du complexe E2F/DP. Par la suite, le facteur de transcription E2F libre peut aller transcrire d'autres gènes responsables de l'avancement subséquent du cycle cellulaire à la phase S. [4], [2]
Régulation de la fonction biologique
Sous l'influence de facteurs mitogènes (par exemple PDGF) les cellules parviennent à lever le frein biologique posé par la présence de pRB hypophosphorylé et ainsi continuer la progression du cycle cellulaire vers la mitose. [5]
Suite à la liaison du facteur mitogène et de son ligand, une cascade de signalisation est enclenchée. Cette cascade de signalisation utilise des kinases pour phosphoryler le substrat suivant de la cascade jusqu'à l'activation du facteur de transcription. Une fois le facteur de transcription activé, cela enclenche la production de protéines appelées cyclines et CDKs, nécessaires pour démarrer le cycle cellulaire. [6]
Les cyclines et CDKs indispensable pour la phase G1, ce qui veux dire qu'ils sont essentiels pour arriver à la phase S, sont les cyclines D et E et les CDKs 2/4/6. Cependant, la cycline D et CDKs 4 et 6 sont produite et activées au tout début de la phase G1, mais ne sont pas capable d'induire la libération du complexe E2F/DP. En effet, on doit attendre à la fin de la phase G1 pour voir la production de cycline E activer CDK 2 qui, à son tour phosphorylera le domaine carboxyl-terminal de pRB. Cette phosphorylation en chaîne par les trois CDKs entraîne un changement de conformation dans la structure de pRB, ce qui l'empêche alors de lier le complexe E2F/DP. Ce complexe étant désormais libre, le facteur de transcription E2F peut alors aller initier la transcription d'ARNm nécessaires à la phase S du cycle cellulaire. [7]
Cependant, il peut aussi y avoir une régulation négative de pRB. En effet, s'il y a des dommages à l'ADN quand la cellule veut entrer en mitose, la protéine p53 se trouve phosphorylée et induit la production de protéine p21. p21 se lie aux complexes cycline/CDK de la phase G1, inhibant ainsi leur activité kinase pour pRB. Il y a par conséquent inhibition de la libération du complexe E2F/DP. [8]
Interactions avec des protéines virales
La structure du domaine B de pRB est hautement conservée à travers la famille de pRB. En effet, plusieurs protéines virales et cellulaires peuvent s'y fixer solidement. Le point commun de ces protéines, virales ou non, se trouve dans une séquence peptidique de neuf acides aminés comportant le motif L (…) C (…) E. [2]
On retrouve spécifiquement ce motif dans les protéines virales E7 des papillomavirus humains. En effet, on retrouve une certaine homologie entre les protéines E7 de chaque souche de VPH. Cependant, certaines études ont démontré que la sévérité oncogénique des souches change selon la force avec laquelle la protéine E7 est liée à pRB.
Ce qui rend les protéines virales E7 oncogènes est leur capacité à se lier sur pRB, libérer par la même occasion le complexe E2F/DP qui fait avancer le cycle cellulaire et accélérer la dégradation de pRB par le protéasome.
L'endroit où se lie l'oncoprotéine E7 sur pRB est connu. En effet, elle se lie à une séquence bien conservée dans le domaine B. Cependant, puisque le site de liaison de l'oncoprotéine se trouve dans le domaine B et que le site de liaison du complexe E2F/DP se trouve dans le domaine C, la raison pour laquelle E7 entraîne la libération de E2F/DP reste toujours énigmatique. [9]
Une fois le complexe E2F/DP relâché, la protéine E7 accélère la dégradation de pRB via la voie du protéasome. Pour y arriver, elle lie et active une enzyme ubiquitaire du cytosol et du noyau, la μ-calpain. La famille des calpains est une famille de protéases de cystéines calcium dépendantes. Une fois activée et liée la μ-calpain ampute pRB de 110 acides aminés à son extrémité carboxyl-terminale. Comme les fonctions de séquestration et de phosphorylation de pRB s'effectuent dans le domaine C, la protéine devient alors incapable de lier E2F et devient par conséquent inutilisable. Les protéines devenues inutilisables dans les cellules sont alors dégradées par le protéasome via la voie de l'ubiquitinisation. [10]
Pathologies souvent associées
- Rétinoblastome (sporadique unilatéral ou héréditaire bilatéral) ;
- Tumeur de Wilms ;
- Ostéosarcome ;
- Cancer de la vessie.
Notes et références
- ↑ Structure
- http ://www. ebi. ac. uk/interpro/IEntry?ac=IPR015652
- ↑ Abouzeid H, Pourvuer F. L, Thonney F, Schorderet D. F (2007) Ten novel RB1 gene mutations in patients with retinoblastoma. Molecular vision. 2007; 13 :1740-5; PMID 17960112.
- ↑ Griffiths, Wessler, Lewontin, Gelbart, Suzuki, Miller. (2006). Introduction à l'analyse génétique. Ch. 17, La régulation génétique du nombre de cellules : les cellules normales et les cellules cancéreuses. P. 549-550 et 564-565.
- ↑ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P. (2004). Essential cell biology. Ch. 18, Cell-cycle control and cell death. P. 629-631
- ↑ Griffiths, Wessler, Lewontin, Gelbart, Suzuki, Miller. (2006). Introduction à l'analyse génétique. Ch. 17, La régulation génétique du nombre de cellules : les cellules normales et les cellules cancéreuses. P. 558.
- ↑ OMIM : http ://www. ncbi. nlm. nih. gov/entrez/dispomim. cgi?id=180200
- ↑ Griffiths, Wessler, Lewontin, Gelbart, Suzuki, Miller. (2006). Introduction à l'analyse génétique. Ch. 17, La régulation génétique du nombre de cellules : les cellules normales et les cellules cancéreuses. P. 550.
- ↑ Mũnger K, Werness B. A, Dyson N, Phelps W. C, Harlow E, Howley P. M. (1989) Complex formation of human papillomavirus E7 proteins with retinoblastoma tumor suppresor gene product. The EMBO journal. 1989; vol. 8 no. 13 pp. 4099-4105; PMID : 2556261
- ↑ Darnell GA, Schroder WA, Antalis TM, Lambley E, Major L, Gardner J, Birrell G, Cid-Arregui A, Suhrbier, A. (2007) Human papillomavirus E7requires the protease calpain to degrade the retinoblastoma protein. J. Biol. Chem.. 2007; PMID : 17977825.
Recherche sur Google Images : |
|
"protéine du rétinoblastome" L'image ci-contre est extraite du site unilim.fr Il est possible que cette image soit réduite par rapport à l'originale. Elle est peut-être protégée par des droits d'auteur. Voir l'image en taille réelle (550 x 575 - 32 ko - gif)Refaire la recherche sur Google Images |
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 05/11/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité