Insertion cotraductionnelle des protéines membranaires
L'insertion cotraductionnelle des protéines membranaires permet l'insertion d'une protéine dans une membrane au cours de sa traduction.
L'insertion cotraductionnelle des protéines membranaires permet l'insertion d'une protéine dans une membrane au cours de sa traduction. Ce processus permet seulement à la formation de protéines à hélices α transmembranaires. L'insertion membranaire des protéines monotopiques ou des protéines à tonneau β transmembranaire se fait par des mécanismes post-traductionnels, de même que pour certaines protéines à hélices α transmembranaires.
Insertion cotraductionnelle des protéines membranaires de type I
Étape 1
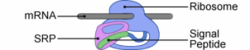
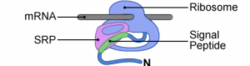
L'insertion cotraductionnelle se déroule à proximité de la membrane du Réticulum Endoplasmique (RE) chez les eucaryotes (de la membrane plasmique chez les procaryotes). La traduction des protéines membranaires de type I débute dans le cytosol par une séquence N-terminale spécifique nommée Peptide Signal (PS). Le PS s'est vu consacré par SRP (Signal Recognition Particule) dès sa sortie du ribosome. SRP se fixe au PS et au ribosome ralentissant, ou alors stoppant, la traduction.
Étape 2
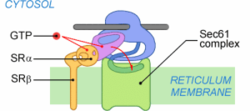
Une fois le PS reconnu, la protéine est adressée à la membrane. Chez les mammifères, SRP s'est vu consacrée par SR (SRP Receptor), un récepteur membranaire du réticulum. L'interaction SRP-SR est régulée par le GTP. La fixation de GTP sur SRP54 permet la dissociation de SRP et du PS[1]. La fixation d'un GTP sur la sous-unité SRα stabilise l'ancrage SRP-SR. La déstabilisation du complexe SRP-PS est compensée par l'ancrage du ribosome au complexe de translocation, ou translocon.
Étape 3
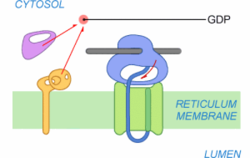
Le transfert à travers la membrane du RE débute. Tout d'abord, le peptide signal s'est vu consacré par le translocon et commence à pénétrer dans le canal [2]. Le complexe SRP-SR se dissocie du ribosome. SRP se dissocie de SR après hydrolyse du GTP en GDP et devient à nouveau opérationnel. Le ribosome n'est plus bloqué et reprend la traduction de l'ARNm. Le peptide en cours de synthèse se retrouve directement orienté vers le canal du translocon.
Étape 4
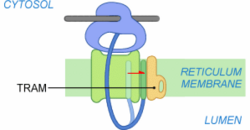
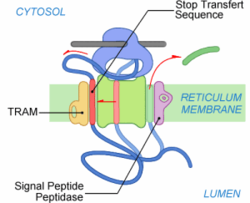
À ce stade, le ribosome est fortement lié au translocon. Le peptide signal se retrouve en contact avec le translocon et TRAM (Translocating Chain Associating Membrane protein), une glycoprotéine satellite du complexe de translocation. TRAM n'est pas toujours requise lors de la translocation. Elle semble tout de même fréquemment indispensable à l'insertion des protéines membranaires. Le contact entre le PS et TRAM est envisageable grâce à une ouverture du canal constitué par Sec61 sur la périphérie du translocon.
Étape 5
La synthèse du polypeptide continue. Il est transféré à travers la membrane au fur et à mesure de son élongation jusqu'à la synthèse d'une séquence hydrophobe spécifique qui bloque le transfert du polypeptide. Cette séquence est nommée Signal d'Arrêt de Transfert (SAT). Le SAT se compose d'une vingtaine de résidus hydrophobes. Grâce à son hydrophobie il suit le même chemin que le peptide signal et se retrouve en périphérie du complexe de translocation, certainement en contact avec TRAM[3], [4]. Cette translation est envisageable grâce à une ouverture latérale du translocon.
Dans un même temps, le PS est clivé dès que le site de clivage est accessible à la Signal Peptidase (SPase). Le site de clivage est localisé en C-terminal du PS. Le PS clivé est ensuite dégradé. On retrouve ses reliquats dans le cytosol. Pendant ce temps, la traduction continue. Le ribosome est toujours lié au translocon, mais la partie C-terminale en cours de synthèse n'est plus transférée à travers le translocon[5].
Étape 6
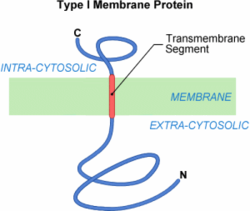
La traduction de la protéine membranaire de type I est achevée. La protéine a directement été insérée avec la bonne topologie membranaire, c'est-à-dire avec une partie N-terminal extra-cytosolique et une partie C-terminale intracytosolique. Cette insertion a emprunté la voie d'exportation des protéines sécrétées dans le milieu extérieure. Elle a obligation en plus une séquence hydrophobe, la SAT, donnant la possibilité l'insertion de la protéine dans le milieu membranaire. La SAT forme par conséquent l'ancrage membranaire de la protéine. L'insertion cotraductionnelle peut finalement être reconnue comme un transfert partiel. Mais il faut savoir que le translocon possède d'autres capacités. Il peut, par exemple, fonctionner dans le sens inverse et renvoyer des protéines en cours de transfert vers le cytosol. On parle alors de retrotranslocation[6]. La translocation cotraductionnelle est par conséquent un mécanisme particulièrement souple et probablement particulièrement finement régulé par la cellule.
Insertion cotraductionnelle des protéines membranaires de type II et III
Peptide signal de type II et III
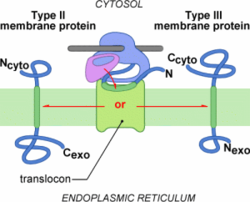
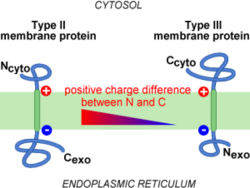
L'insertion cotraductionelle des protéines membranaires de type II et III utilise aussi le complexe de translocation. C'est le peptide signal qui est différent car il est responsable à la fois de l'adressage au translocon et de l'ancrage membranaire. Ce Peptide Signal ne possède pas de site de clivage comme dans les cas des protéines de type I. Il est le plus souvent plus long (de 18 à 25 résidus principalement apolaires) dans la mesure où il doit être suffisamment long pour former une hélice transmembranaire[7]. De plus, le peptide signal des protéines membranaires de type II et III est situé au sein de la séquence plutôt qu'à l'extrémité N-terminale. On parle aussi de Signal d'Ancrage pour désigner le Peptide Signal des protéines de type II et III.
Mais quelle est la différence entre les protéines membranaires de type II et de type III ? En réalité, le signal d'ancrage de type II induit une translocation C-terminale, comme dans le cas du peptide signal de type I. Par contre, le signal d'ancrage de type III induit une translocation N-terminale. On parle alors de signal d'ancrage reverse.
Déterminants de l'orientation du signal d'ancrage
Règle du positif à l'intérieur
Le déterminant le mieux connu de l'orientation du Signal d'Ancrage est la distribution des charges à proximité immédiate du cœur hydrophobe du signal. C'est la règle du positive inside (positif à l'intérieur). C'est à dire, les résidus basiques sont statistiquement quatre fois plus abondants du côté cytoplasmique que du côté périplasmique chez les bactéries[8]. La corrélation inverse existe aussi pour les résidus acides (plus de résidus acides dans les segments périsplasmiques), quoique moins marquée. La règle du «positive-inside» se vérifie aussi avec les protéines membranaire du Réticulum Endoplasmique, du chloroplaste et de la mitochondrie[9], [10].
Dans le cas de l'insertion cotraductionnelle, c'est plutôt la différence de charge de part et d'autre du cœur hydrophobe qui guide l'orientation de l'insertion. C'est le côté le plus positif qui est le plus souvent cytoplasmique[11]. Il est par conséquent envisageable l'orientation du signal en modifiant Ses charges flanquantes[12], [13]. Le changement d'orientation ne touche le plus fréquemment qu'une partie des protéines produites. Cela veut dire tout simplement que la distribution des charges n'est pas l'unique déterminant de la topologie membranaire. positive inside rule
Quels sont les facteurs responsables de la règle du «positive inside» ? La réponse n'est pas encore clairement établie à l'heure actuelle. Il est certain que les phospholipides ont une influence importante sur la translocation mais il est complexe de connaître sont rôle exact dans l'orientation des peptides lors de la translocation. Il a été démontré que les phospholipides anioniques pouvaient retenir les charges positives des peptides signaux procaryotes et empêcher leur translocation, suggérant une interaction peptide-lipide directe[14]. Des facteurs protéines interviennent certainement aussi. Il y a peu de temps, il a été démontré que Sec61p (chez Saccharomyces cerevisiæ) contribuait à l'orientation du peptide signal certainement par des interactions de type électrostatique[15]. Il semble que les charges positives puissent directement avec les phospholipides chargés négativement dans le cas des procaryotes[14].
Hydrophobie du signal
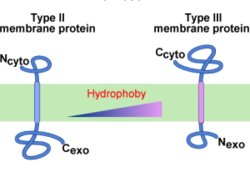
Généralement, plus le signal d'ancrage est hydrophobe, plus la translocation est N-terminale, i. e. l'insertion est de type III[16]. Un signal d'ancrage suffisamment hydrophobe peut même outrepasser la règle du «positive inside»[17].
Comment l'hydrophobie du signal intervient dans la topologie membranaire ? Il a été proposé que ce soit un gradient apolaire, plutôt qu'une hydrophobie globale, qui intervienne dans l'orientation du signal[18]. Apparemment, un signal plus hydrophobe sur la fin est plus efficacement transloqué. Cela peut suggérer l'existence d'un site d'ancrage du signal dans le translocon plus hydrophobe du côté extracytoplasmique[16].
Repliement du domaine N-terminal
Dans le cas des protéines de type II et III, un segment N-terminal est le plus souvent synthétisé avant le peptide signal. Le repliement d'un domaine N-terminal peut par conséquent interférer avec la translocation à travers le translocon et faciliter la rétention de la partie N-terminale dans le cytosol[19]. Ainsi, énormément de protéines de type III n'ont pas réellement de domaine structural en N-terminal pour favoriser la translocation. Dans les autres cas, il est envisageable que des chaperons interviennent pour empêcher un repliement stable.
Références
- ↑ (en) Miller JD, Wilhelm H, Gierasch L, Gilmore R, Walter P. (1993) GTP binding and hydrolysis by the signal recognition particle during initiation of protein translocation, Nature, vol. 366 :351-354 pp.
- ↑ (en) Mothes W, Jungnickel B, Brunner J, Rapoport TA. (1998) Signal sequence recognition in cotranslational translocation by protein components of the endoplasmic reticulum membrane, J. Cell Biol. , vol. 142 :355-364 pp.
- ↑ (en) Mothes W, Heinrich SU, Graf R, Nilsson I, von Heijne G, Brunner J, Rapoport TA (1997) Molecular mechanism of membraneprotein integration into the endoplasmic reticulum, Cell, vol. 89 (4) :523-533 pp.
- ↑ (en) Do H, Falcone D, Lin J, Andrews DW, Johnson Æ. (1996) The cotranslational integration of membrane proteins into the phospholipid bilayer is a multistep process, Cell, vol. 85 :369-378 pp.
- ↑ (en) Heinrich SU, Mothes W, Brunner J, Rapoport TA. (2000) The Sec61p complex mediates the integration of a membrane protein by allowing lipid partitioning of the transmembrane domain, Cell, vol. 102 :233-244 pp.
- ↑ Johnson Æ, Haigh NG. (2000) The ER translocon and retrotranslocation : is the shift into reverse manual or automatic?, Cell, vol. 102 :709-712 pp.
- ↑ (en) Goder, V., Spiess, M. (2001) Topogenesis of membrane proteins : determinants and dynamics, FEBS Lett. , vol. 504 :87-93 pp.
- ↑ (en) von Heijne, G. (1986) Net N-C charge imbalance may be important for signal sequence function in bacteria, J. Mol. Biol. , vol. 192 :287-290 pp.
- ↑ (en) von Heijne, G. (1992) Membrane protein structure prediction. Hydrophobicity analysis and the positive-inside rule, J. Mol. Biol. , vol. 225 :487-494 pp.
- ↑ (en) Wallin, E., von Heijne, G. (1998) Genome-wide analysis of integral membrane proteins from eubacterial, archæan, and eukaryotic organisms, Protein Sci. , vol. 7 :1029-1038 pp.
- ↑ (en) Hartmann, E., Rapoport, T. A., Lodish, H. F. (1989) Predicting the orientation of eukaryotic membrane-spanning proteins, Proc. Natl. Acad. Sci. USA, vol. 86 :5786-5790 pp.
- ↑ (en) Parks GD, Lamb RA. (1993) Role of NH2-terminal positively charged residues in establishing membrane protein topology, J. Biol. Chem. , vol. 268 :19101-19109 pp.
- ↑ (en) Rutz, C., Rosenthal W., Schulein, R. (1999) A single negatively charged residue affects the orientation of a membrane protein in the inner membrane of Escherichia coli only when it is located adjacent to a transmembrane domain., J. Biol. Chem. , vol. 274 :33757-33763.
- (en) van Klompenburg W, Nilsson I, von Heijne G, de Kruijff B. (1998) Anionic phospholipids are determinants of membrane protein topology, EMBO J. , vol. 16 :4261-4266.
- ↑ (en) Goder, V., Junne, T., Spiess, M. (2003) Sec61p contributes to signal sequence orientation according to the positive-inside rule, Mol. Biol. Cell. , vol. 15 :1470-1478 pp.
- (en) Goder, V., Spiess, M. (2003) Molecular mechanism of signal sequence orientation in the endoplasmic reticulum, EMBO J. , vol. 22 :3645-3653 pp.
- ↑ (en) Rosch, K., Næher, D., Laird, V., Goder, V., Spiess, M. (2000) The topogenic contribution of uncharged amino acids on signal sequence orientation in the endoplasmic reticulum, J. Biol. Chem. , vol. 275 :14916-14922 pp.
- ↑ (en) Harley, C. A., Holt, J. A., Turner, R., Tipper, D. J. (1998) Transmembrane protein insertion orientation in yeast depends on the charge difference across transmembrane segments, their total hydrophobicity, and its distribution, J. Biol. Chem. , vol. 273 :24963-24971 pp.
- ↑ (en) Denzer, A. J., Nabholz, C. E., Spiess M. (1995) Transmembrane orientation of signal-anchor proteins is affected by the folding state but not the size of the N-terminal domain, EMBO J. , 'vol. 14 :6311-6317 pp.
Recherche sur Google Images : |
|
"sur/Insertion" L'image ci-contre est extraite du site fr.wikipedia.org Il est possible que cette image soit réduite par rapport à l'originale. Elle est peut-être protégée par des droits d'auteur. Voir l'image en taille réelle (200 x 162 - 26 ko - )Refaire la recherche sur Google Images |
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 05/11/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité