Séquençage par spectrométrie de masse
Voir aussi Séquençage des protéines
Voir aussi Séquençage des protéines
La spectrométrie de masse MS/MS sert à déterminer la séquence des peptides sur quelques dizaine d'acides aminés. Couplé avec des techniques de digestion (enzymatiques ou chimiques) et avec des techniques de séparation (chromatographie en phase liquide à haute performance par exemple), il est envisageable de caractériser une protéine.
Principe de la fragmentation
La fragmentation est le processus servant à séparer chaque acide aminé d'un peptide. Pour des raison de stabilité des liaisons, les séquences polypeptidiques se clivent préférentiellement au niveau des liaisons peptidiques. Il existe cependant des mécanismes complexes qui produisent d'autres types ruptures de liaison, ce qui peut rendre complexe la lecture des spectres de masses et la détermination de la séquence.
- Spectrométrie de masse MS/MS
Pour fragmenter les peptides, on utilise soit plusieurs analyseurs couplés en série (spectromètre de type triple quadripoles QqQ ou de type quadripole-temps de vol Q-TOF) ou un analyseur capable de réaliser plusieurs analyses en série (spectromètre de masse de type trappe ionique IT ou de type résonance cyclotronique FT-ICR).
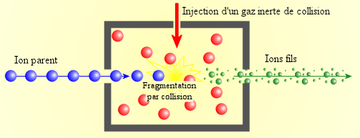
- Le premier analyseur sert à sélectionner un ion à fragmenter (appelé "ion parent") et d'exclure l'ensemble des autres ions présents.
- Cet ion parent est fragmenté, généralement par collision avec un gaz inerte (c'est-à-dire qui ne réagit pas chimiquement avec l'ion parent) dans une cellule de collision. Cette fragmentation sert à "casser" les liaisons moléculaires, surtout les liaisons peptidiques, et de produire des "ions fils".
- Les ions fils sont ensuite scindés par le second analyseur puis détectés.
- Mode d'analyse envisageable
Il existe plusieurs mode d'analyse en spectrométrie de masse en tandem :
- Fragment ion scan (ou Product ion scan' ou Daughter scan)
- Dans ce mode, un ion parent de rapport m/z donné est choisi. Il est fragmenté puis la totalité de la gamme de masse est balayée pour analysé les ions fils produits.
- Precursor ion scan (ou Parent scan)
- Dans ce mode, un seul ion fils de rapport m/z donné est analysé. La totalité de la gamme de masse des ions parents est balayé. Ce mode permet d'analyser les ion parents qui produisent particulièrement un ion fils donné.
- Neutral ion scan
- Dans ce mode, les ions parents et les ions fils sont analysés sur la totalité de la gamme de masse. On choisit un masse m1, on balaye les ions parents sur la totalité de la gamme de masse et on analyse les ions fils produits de masse m2-m1. Ce mode permet d'étudier les ions parents qui peuvent perdre un fragment neutre (et qui ne peut par conséquent pas être analaysé par le second balayage). A titre d'exemple, on peut analysé les alcools en effectuant recherchant une différence de masse de 18 Da.
- Sans balayage
- Dans ce mode, la masse de l'ion parent et de l'ion fils est déterminé. Ce mode est utilisé en quantification pour mesurer la quantité d'une molécule donnée.
- Type de fragmentation
Les fragmentations peuvent être classé selon leurs types :
- Fragmentation spontanée des ions métastables ou fragmentation induite par collision sur une surface (SID Surface Induced Dissociation), avec un gaz inerte (CID Collision Induced Dissociation) ou un gaz réactif (RIM Reactant Ion Monitoring)
- Fragmentation de haute énergie (par exemple avec un TOF/TOF), produisant énormément de rupture de liaisons, ou fragmentation de basse énergie, minimisant les ruptures de liaisons non peptidiques.
- Stabilité des ions
En fonction de l'énergie interne des ions, les ions se fragmentent à différents temps de vie :
- ion stable : plus de 10-6 s
- ion métastable : entre 10-6 et 10-7 s
- ion instable : moins de 10-7 s
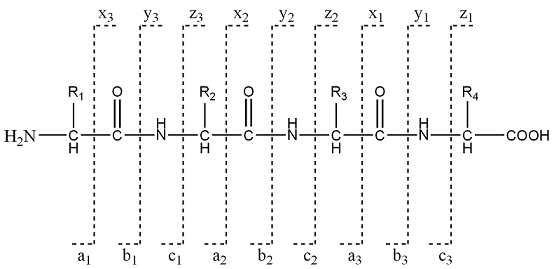
Série de fragmentation
M = M(n AA) + M(H) + M(OH)
| Code à 1 lettre | Code à 3 lettres | Acide aminé | Masses | Acides aminés modifiés |
| A | Ala | Alanyl | 71 | |
| C | Cys | Cystyl | 103 | |
| D | Asp | Aspartyl | 115 | |
| E | Glu | Glutamyl | 129 | |
| F | Phe | Phénylalanyl | 147 | - |
| G | Gly | Glycyl | 57 | - |
| H | His | Histidyl | 137 | 153 (Oxydation) |
| I | Ile | Isoleucyl | 113 | - |
| K | Lys | Lysyl | 128 | 255 (SMA) |
| L | Leu | Leucyl | 113 | - |
| M | Met | Méthionyl | 131 | 147 (Oxydation) 163 (Sulfonation) |
| N | Asn | Asparagyl | 114 | - |
| P | Pro | Prolyl | 97 | - |
| Q | Gln | Glutamyl | 128 | |
| R | Arg | Arginyl | 156 | - |
| S | Ser | Séryl | 87 | 167 (Phosphorylation) |
| T | Thr | Thréonyl | 101 | 181 (Phosphorylation) |
| V | Val | Valyl | 99 | - |
| W | Trp | Tryptophanyl | 186 | 206 (Oxydation) |
| Y | Tyr | Tyrosyl | 163 | 243 (Phosphorylation) |
| Modification | Masse |
| Acétylation | + 42 Da |
| + 226 Da | |
| + 43 Da | |
| + 28 Da | |
| SMA | + 127 Da |
| Modification | Masse |
| + 14 Da | |
| + 22 Da |
Fragmentation au niveau de la liaison peptidique
Fragmentation d'une seule liaison
Fragmentation N terminale
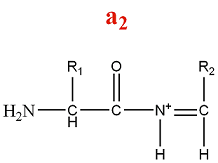
- Série d'ions a
La fragmentation de la série a produit des ions aldimines :
M = M(n AA) + M(H) - M(CO) = M(n AA) - 27
La série a* dérive de la série a par la perte d'un groupe NH3 :
M = a - M(NH3) = a - 17
La série a0 dérive de la série a par la perte d'un groupe H2O :
M = a - M(H2O) = a - 18
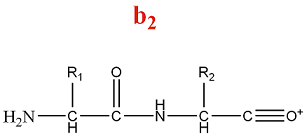
- Série d'ions b
La fragmentation de la série b produit des ions acylium :
M = M(n AA) + M(H) = M(n AA) + 1
La série b* dérive de la série b par la perte d'un groupe NH3 :
M = b - M(NH3) = b - 17
La série b0 dérive de la série b par la perte d'un groupe H2O :
M = b - M(H2O) = b - 18
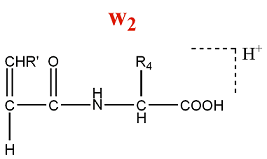
- Extrémité N terminale
Le fragment peptidique qui plus est faible poids moléculaire de la série b correspond à l'extrémité N terminal du peptide parent. Il correspond à la formule :
R
NH2-CH-CO+
et il donne un pic avec un rapport m/z :
m/z = M(H) + M(aa)
= M(aa) + 1
- Extrémité C terminale
Le fragment peptidique qui plus est haut poids moléculaire de la série b correspond à l'extrémité C terminal du peptide parent [M+H]+. Il correspond à la formule :
R
NH2-...-(CH)-...-CO+
La différence de masse entre ce fragment et le peptide parent correspond par conséquent au dernier acide aminé aan :
m/z = M(ion parent M[M+H]+) - M(fragment peptidique)
= ( M(H) + SM(aa) + M(aan + M(OH) + M(H+) ) - ( M(H) + SM(aa) )
= M(aan ) + 18
En particulier, dans le cas d'une digestion trypsique, l'acide aminé C terminal correspond à une lysine ou une arginine. Selon cet acide aminé, on observe un pic qui apparait aux différence suivante comparé au rapport m/z de l'ion parent [M+H]+ :
m/z = 146 pour la lysine
et
m/z = 174 pour l'arginine
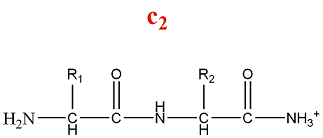
- Série d'ions c
La fragmentation de la série c produit des ions amino :
M = M(n AA) + M(H) + M(NH3) = M(n AA) + 18
Fragmentation C terminale
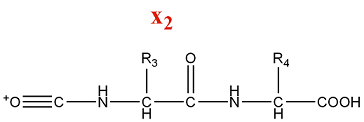
- Série d'ions x
La fragmentation de la série x produit des ions acylium :
M = M(n AA) + M(CO) + M(OH) = M(n AA) + 45
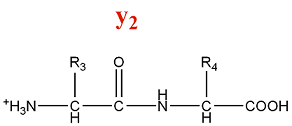
- Série d'ions y
La fragmentation de la série y produit des ions amino :
M = M(n AA) + M(2H) + M(OH) = M(n AA) + 19
La série y* dérive de la série y par la perte d'un groupe NH3 :
M = y - M(NH3) = y - 17
La série y0 dérive de la série y par la perte d'un groupe H2O :
M = y - M(H2O) = y - 18
- Extrémité C terminale
Le fragment peptidique qui plus est faible poids moléculaire de la série y correspond à l'extrémité C terminal du peptide parent. Il correspond à la formule :
R
NH3+-CH-COOH
et il donne un pic avec un rapport m/z :
m/z = 2M(H) + M(aa) + M(OH)
= M(aa) + 19
En particulier, dans le cas d'une digestion trypsique, l'acide aminé C terminal correspond à une lysine ou une arginine. Selon cet acide aminé, on observe un pic aux valeurs :
m/z = 147 pour la lysine
et
m/z = 175 pour l'arginine
- Extrémité N terminale
Le fragment peptidique qui plus est haut poids moléculaire de la série y correspond à l'extrémité N terminal du peptide parent. Il correspond à la formule :
R
NH3+-...-(CH)-...-COOH
La différence de masse entre ce fragment et l'ion parent [M+H]+ correspond par conséquent au premier acide aminé aa1 :
m/z = M(ion parent M[M+H]+) - M(fragment peptidique)
= ( M(H) + SM(aa) + M(aa1 + M(OH) + M(H+) ) - ( 2M(H) + SM(aa) + M(OH) )
= M(aa1 )
- Série d'ions z
- La fragmentation de la série z produit des carbocations :
M = M(n AA) - M(NH) + M(OH) = M(n AA) + 2
Fragmentation de deux liaisons
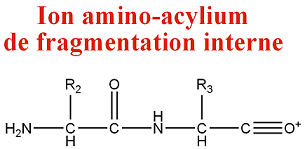
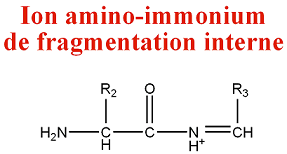
- Ion de fragmentation interne
Ces ions correspondent au clivage de deux liaisons peptidiques. Ces clivages correspondent généralement à des fragmentations de types b et y et forment des ions amino-acylium :
M = M(n AA) + M(2H) = M(n AA) + 2
Quelquefois, les clivages correspondent à des fragmentations de types a et y et produisent des ions amino-immonium :
M = M(n AA) + M(H) - M(CO) = M(n AA) - 27
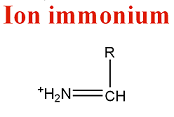
- Ion immonium
Quand qu'une fragmentation de type a est voisine d'une fragmentation de type y, il se forme un ion immonium :
M = M(AA) + M(H) - M(CO) = M(AA) - 27
Les ions immoniums apparaissent dans un spectre MS/MS à des masses inférieurs à 200 Da. Ils permettent de connaître la composition en acide aminé du peptide fragmenté.
| Acide aminé | Code à 3 lettres | Code à 1 lettres | Masse | Ion immonium dérivé |
| Acide aspartique | Asp | D | 88 | 70 |
| Acide glutamique | Glu | E | 102 | - |
| Alanine | Ala | A | 44 | - |
| Arginine | Arg | R | 129 | 59, 70, 73, 87, 100, 112 |
| Asparagine | Asn | N | 87 | 70 |
| Cystéine | Cys | C | 76 | - |
| Glutamine | Gln | Q | 101 | 56, 84, 129 |
| Glycine | Gly | G | 30 | - |
| Histidine | His | H | 110 | 82, 121, 123, 138, 166 |
| Isoleucine | Iso | I | 86 | 44, 72 |
| Leucine | Leu | L | 86 | 44, 72 |
| Lysine | Lys | K | 101 | 70, 84, 112, 129 |
| Méthionine | Met | M | 104 | 61 |
| Phénylalanine | Phe | F | 120 | 91 |
| Proline | Pro | P | 70 | - |
| Sérine | Ser | S | 60 | - |
| Thréonine | Thr | T | 74 | - |
| Tryptophane | Trp | W | 159 | 77, 117, 130, 132, 170, 171 |
| Tyrosine | Tyr | Y | 136 | 91, 107 |
| Valine | Val | V | 72 | 41, 55, 69 |
Fragmentation des chaînes latérales
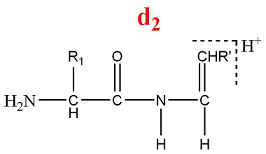
- Série d'ions d
Un ion de la série d est constitué par la perte d'une partie de la chaîne latérale de l'acide aminé C terminal d'un ion de la série a.
M = M(AA) + M(H) - M(CO) - M(Rperdu) = M(AA) - 27 - M(Rperdu)
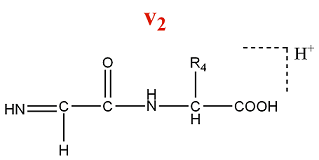
- Série d'ions v
Un ion de la série v est constitué par la perte de la chaîne latérale de l'acide aminé N terminal d'un ion de la série y.
M = M(AA) + M(OH) - M(Rperdu) = M(AA) + 17 - M(Rperdu)
- Série d'ions w
Un ion de la série w est constitué par la perte d'une partie de la chaîne latérale de l'acide aminé N terminal d'un ion de la série z.
M = M(AA) + M(OH) - M(NH) - M(Rperdu) = M(AA) + 2 - M(Rperdu)
Liste des modifications des protéines
Recherche sur Google Images : |
|
"Un article de Wikipédia," L'image ci-contre est extraite du site fr.wikipedia.org Il est possible que cette image soit réduite par rapport à l'originale. Elle est peut-être protégée par des droits d'auteur. Voir l'image en taille réelle (554 x 269 - 30 ko - )Refaire la recherche sur Google Images |
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 05/11/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité